如何获取Ensembl gene id和NCBI的gene id及与HGNC的对应关系
Ensembl和NCBI都是盛名的基因组研究机构,提供相关的基因组结构注释文件,比如gtf或者gff,但注释的id却不是统一的。比如基因ID,Ensembl有Ensembl gene id,NCBI有entrez gene id。不同的人用的基因注释文件来源不同,就需要进行转换。本文主要讲如何利用Ensembl的Biomart,下载对应关系。
Biomart整合了各种生物学注释数据,提供了易于操作的界面,在线提供批量下载,以加速科学研究。Ensembl已应用biomart提供相关服务。
The BioMart project provides free software and data services to the international scientific community in order to foster scientific collaboration and facilitate the scientific discovery process. The project adheres to the open source philosophy that promotes collaboration and code reuse.
Ensembl的biomart网址为http://asia.ensembl.org/index.html
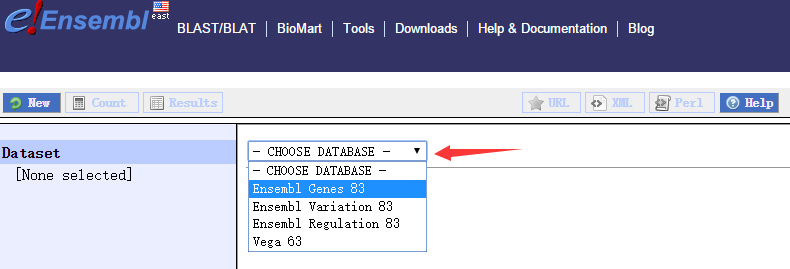
第一步,选择相应的数据库
选择ensemble gene 83
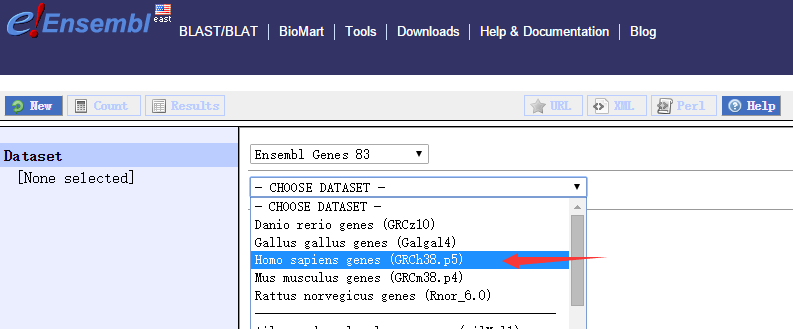
选择homo sapiens gene

 )
)
