AMY-Tree分析Y染色体 ---分类树
AMY-Tree下载地址
https://bio.kuleuven.be/eeb/lbeg/software 解压下载的压缩文件
AMY-Tree输入文件
输入文件的格式要求在解压出来的 Manual (AMY-tree v2.0 User Manual.pdf)里可以看到

https://bio.kuleuven.be/eeb/lbeg/software 解压下载的压缩文件
输入文件的格式要求在解压出来的 Manual (AMY-tree v2.0 User Manual.pdf)里可以看到
breakdancer在call structure variant的时候,生成的文件中只有一行报错的信息 Error: no bams files in config file!
这是因为breakdancer在call SV之前,要先生成配置文件cfg文件。生成cfg文件的依据,是统计bam文件中paired read的插入长度,read的平均长度等信息。如果bam文件中的read都不是配对的或者很少有配对的,在运行bam2cfg.pl时,就会生成空的配置文件,导致检测不出SV,报 Error: no bams files in config file! 错误。
解决方法:
1,准备正确的bam文件,确保足够的配对读长paired reads,以便生成cfg配置文件
2,同一批样本的插入长度,read平均长度等都差不多,可以将其他文件生成的cfg文件中的 map: 选项后改为你的bam文件。即利用其他bam文件生成的cfg文件,当作要检测SV的bam的配置。
在1000genome的FTP服务器上可以下载一个all的vcf文件,里面可以看到AFR, AMR, EAS, EUR, SAS人群的allele频率,但是该种族下面的亚群的频率信息需要在http://grch37.ensembl.org搜索得到,比如 http://grch37.ensembl.org/Homo_sapiens/Variation/Population?db=core;r=1:230845294-230846294;v=rs699;vdb=variation;vf=102788013 ,还有一种方式,就是下载包含所有样本的突变信息的VCF文件,利用vcftools计算。
The allele frequency of super population (AFR, AMR, EAS, EUR, SAS, see http://www.1000genomes.org/category/population/) can be obtained from all.vcf.
However, the allele population frequency in subpopulation is not well obtained.
One way is search the web http://grch37.ensembl.org by rs identifier, E.g. http://grch37.ensembl.org/Homo_sapiens/Variation/Population?db=core;r=1:230845294-230846294;v=rs699;vdb=variation;vf=102788013 .
Another way is calculated on your local machine. The following will introduce how to get allele frequency of CHB population in chr1 chromosome. CHB Han Chinese in Beijing
|
|
# . 表示允许所有表 ‘root’ 表示允许root用户 ‘%’ 表示任意ip ‘123456’ 表示密码
|
|
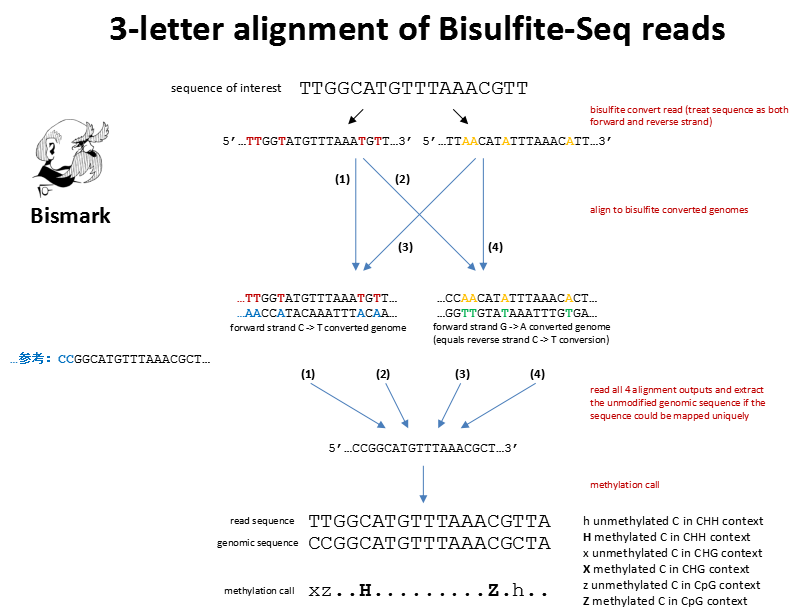
bismark调用bowtie2进行比对,调用samtools生成bam文件,因此在运行bismark之前,需要安装bowtie2和samtools
请注意,fastq文件要进行质控,比如去掉低质量的reads,去掉adaptor等,可以看本文最下方推荐的PPT,本文不介绍,此外本本只介绍到序列比对,后续的统计分析没有介绍,有兴趣的朋友可以关注swDMR和methykit工具包。
|
|
# --path\_to\_bowtie后面跟的是文件夹
# --verbose 输出log信息
# ./ref 文件夹中有一个基因组fasta文件
# --bowtie2指明用的是bowtie2
./bismark\_v0.16.1/bismark\_genome\_preparation --path\_to_bowtie /home/zzx/bowtie2-2.2.9/ --bowtie2 --verbose ./ref/
因为在重亚硫酸盐甲基化测序中,因为未甲基化的C会变为T,在正链表现为C–>T,但是在负链有C变为T,转换为正链时,即为G–>A,所以基因组需要进行两种转化,才能用于比对。在基因组目录下产生Bisulfite_Genome目录,有CT_conversion和GA_conversion文件夹,这两个文件夹包含转换后的fasta文件和bowtie2建立的索引bt2文件。
fastq中的BS转换后的read与转换的参考基因组比对,得到在参考基因组中的位置,再与原始的参考基因组比较,确定methylate call