用SRA-Explorer辅助下载测序数据
下载数据的时候,偶然碰到了SRA-Explorer,感觉挺好用的,地址:https://sra-explorer.info/
这个页面本身非常小,见https://github.com/ewels/sra-explorer,利用的是SRA API。
检索好之后,选择你想下载的样本,点击Add ** to collection,然后点击右上角saved datasets,页面下方就可以原始的fastq的链接,用curl下载fastq的命令,用aspera下载fastq的命令,还有下载SRA的命令,以及样本的metadata。非常好用。

预测模型校准曲线 Calibration curve
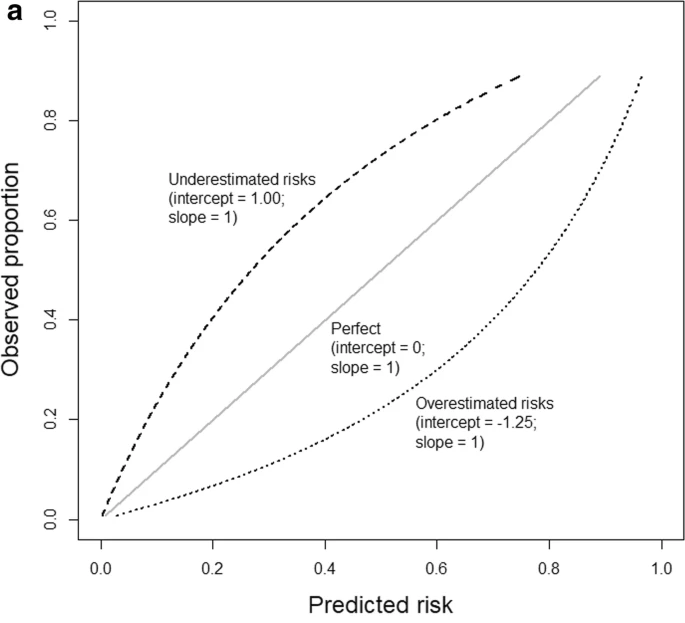
我们常用ROC曲线来衡量模型的预测能力,但很少关注模型的校准度calibration。
Calibration curve的横坐标是我们用模型预测的probability,比如我预测的是可能是肿瘤患者的概率,risk probability,纵坐标是真实的事件event的概率或者事件的proportion。
特征选择
特征选择是机器学习中的一个重要步骤,通过特征选择挑选出对预测起重要作用的变量,既可以减少数据的维度,也可以减少计算的消耗,同时也有助于我们对自己的数据的理解。有许多方法都可以应用到特征选择,比如大家常用的LASSO。我在用R做数据分析的时候,看到过这个帖子,进而了解了很多算法,所以对这个帖子进行了翻译,方便自己复习,也方便大家学习。
原文参考: https://www.machinelearningplus.com/machine-learning/feature-selection/
- Boruta
- Variable Importance from Machine Learning Algorithms
- Lasso Regression
- Step wise Forward and Backward Selection
- Relative Importance from Linear Regression
- Recursive Feature Elimination (RFE)
- Genetic Algorithm
- Simulated Annealing
- Information Value and Weights of Evidence
- DALEX Package
Introduction
真实的数据中,有些变量可能仅是噪声,并没有多少重要意义。
这类变量占用内存空间、消耗计算资源,我们最好去除这类变量,特别是在很大的数据集中。
有时候,我们有一个具有业务意义的变量,但不确定它是否确实有助于预测Y。还有一个事实:在一个机器学习算法中有用的特征(例如决策树) 可能其他算法中(例如回归模型)不被选用或者低估。
同时,有些变量单独预测Y的性能不好,但与其他预测变量/特征组合的情况下却非常显著。比如说有些变量与预测指标的相关性很低,但在其他变量参与的情况下,它可以帮助解释某些其他变量无法解释的模式/现象。
在这些情况下,很难决定包含还是去掉这些变量/特征。
这里讨论的策略可以解决这些问题,同时可以帮助理解对于一个模型而言,变量的重要性与否importance,以及对模型有多少贡献。
重要的一点是,我们最希望使用的变量是既具有业务意义同时也有重要性方面的指标。
我们这里导入Glaucoma 数据集,此数据集的目标是通过63个不同的生理测量指标来预测青光眼的与否。
|
|
将ggplot导出成ppt的R包
1,export的graph2ppt函数
https://github.com/tomwenseleers/export
export虽然从CRAN下架了,但依然可以通过github的库来安装,devtools::install_github(“tomwenseleers/export”)


