分析带UMI标签的测序数据
检测癌组织的低频突变,为了提高检测低频突变的灵敏度,往往进行高深度的测序。但样本之间存在交叉污染,测序有存在一定概率的错误,这些因素会导致高深度测序过程中将假阳性的信号放到,得到假阳性的结果。解决交叉污染的方法,有公司比如IDT采用唯一配对的样本index,只有配对的index中的reads才属于特定样本。解决测序错误的方法,研究人员在建库的时候,先对分子加上UMI碱基,unique molecular identifier -> UMI,然后根据来源于同一个分子的测序数据进行测序错误修正,得到正确的分子序列。两种方法结合可以减少交叉污染提高准确性(https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5759201/)。
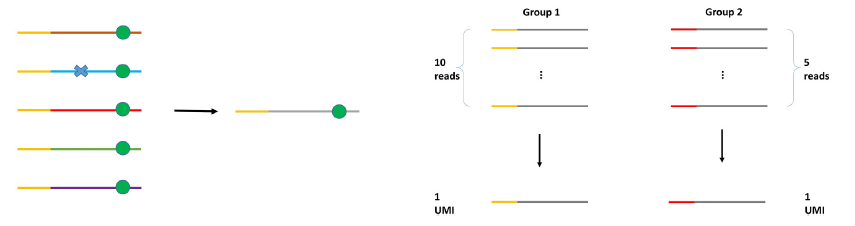
如图中所示,左侧一个分子被测了5次,其中第二次有一个测序错误,但该错误并没有在每个测序数据中出现,所以在后续合成一个分子的时候,测序错误被修正,只保留了真正的突变。(https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5852328/)
常规的肿瘤配对测序分析,或者遗传性突变位点的分析,并不需要UMI信息,所以包含UMI的数据分析是需要不一样的分析流程来得到准确的分析结果,其中包括提取UMI分子标签,合并来自同一个分子的测序reads,低频突变检测而非胚系突变检测等。
大致流程为:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
|
-----prepare analysis ready BAM file------
' FASTQ
' ↓
' uBAM
' ↓ Extract UMI
' uBAM
' ↓ Align uBAM and merge
' BAM
-----call consensus------
' ↓ Group Reads By Umi
' BAM
' ↓ Group Reads By Umi
' BAM
' ↓ Call Molecular Consensus Reads
' uBAM
' ↓ Align uBAM and merge
' BAM
' ↓ Filter Consensus Reads
' BAM
' ↓ Clip
' BAM
-----Vardict------
' ↓ Call
' VCF
|
一,得到包含UMI分子标签信息的BAM文件
UMI信息,应该从fastq中的配对的reads中提取,但fastq不能存储更多的信息,所以需要先将fastq转成uBAM文件,提取uBAM文件中的UMI分子标签信息,将该信息通过RX标签写入uBAM文件中,通过uBAM和BAM文件合并,把RX信息合并到比对到的BAM文件中进行下一步分析。
1)生成uBAM
1
2
3
4
5
6
7
8
9
|
java -Xmx8G -jar picard.jar FastqToSam \
FASTQ=$fq1 \
FASTQ2=$fq2 \
OUTPUT=test.uBAM \
READ_GROUP_NAME=test \
SAMPLE_NAME=test \
LIBRARY_NAME=test \
PLATFORM_UNIT=HiseqX10 PLATFORM=illumina \
RUN_DATE=`date --iso-8601=seconds`
|
2)提取UMI信息
1
2
3
4
5
6
|
java -jar fgbio.jar ExtractUmisFromBAM \
--input=test.uBAM \
--output=test.umi.uBAM \
--read-structure=2M148T 2M148T \
--single-tag=RX \
--molecular-index-tags=ZA ZB
|
此时,uBAM文件中RX标签记录着UMI的信息,2M148T表示前两个碱基是UMI分子标签,148个template 测序序列,配对read在uBAM文件中有如下信息
1
2
|
R1----ZA:Z:TC ZB:Z:TA RG:Z:test RX:Z:TC-TA
R2----ZA:Z:TC ZB:Z:TA RG:Z:test RX:Z:TC-TA
|
3)比对uBAM文件中的reads
1
2
|
samtools fastq test.umi.uBAM ' bwa mem -t 8 -p reference.fa /dev/stdin ' samtools view -b
> test.umi.BAM
|
4)uBAM和BAM合并
1
2
3
4
5
6
7
8
9
10
|
java -Xmx8G -jar picard.jar MergeBAMAlignment R=reference.fa \
UNMAPPED_BAM=test.umi.uBAM \
ALIGNED_BAM=test.umi.BAM \
O=test.umi.merged.BAM \
CREATE_INDEX=true \
MAX_GAPS=-1 \
ALIGNER_PROPER_PAIR_FLAGS=true \
VALIDATION_STRINGENCY=SILENT \
SO=coordinate \
ATTRIBUTES_TO_RETAIN=XS
|
通过合并,得到一个包含各种信息包括RX tag等的BAM文件,该文件用于下一步call consensus read,因为将来源于同一个分子的read合并成一个consensus read,所以在得到BAM文件之后,没有进行mark duplication。
二,Call Consensus Reads
1)Group Reads By Umi
该步会生成一个包含MI tag的文件,存储每个read的最初分子的ID,低质量的read应该过滤掉,因为低比对质量的read上mismatch较多,容易造成假阳性。
1
2
3
4
|
java -jar fgbio.jar GroupReadsByUmi \
--input=test.umi.merged.BAM \
--output=test.umi.group.BAM \
--strategy=paired --min-map-q=20 --edits=1 --raw-tag=RX
|
2)Call Molecular Consensus Reads
根据UMI分子标签的信息和Read1、Read2的位置,从一组read中识别出最初的 molecular 分子序列。min-reads是必填的,如果测序深度较低,单个read也可以用来call consensus,此时设置1,如果深度较高可以设置2或者更高,但1是最安全的,因为后续可以基于此参数进一步过滤。但同时该值的设置显著影响效率,此时我们设置1。此时生成的文件是uBAM格式的,需要进一步比对
1
2
3
4
5
|
java -jar fgbio.jar CallMolecularConsensusReads \
--min-reads=1 \
--min-input-base-quality=20 \
--input=test.umi.group.BAM \
--output=test.consensus.uBAM
|
3)比对uBAM文件中的reads
1
|
samtools fastq $dir/$smp.callconsensus.BAM ' bwa mem -t 8 -p reference.fa /dev/stdin ' samtools view -b - ' test.consensus.BAM
|
4)uBAM和BAM合并
1
2
3
4
5
6
7
8
9
10
|
java -Xmx8G -jar picard.jar MergeBAMAlignment R=reference.fa \
UNMAPPED_BAM=test.consensus.uBAM \
ALIGNED_BAM=test.consensus.BAM \
O=test.consensus.merge.BAM \
CREATE_INDEX=true \
MAX_GAPS=-1 \
ALIGNER_PROPER_PAIR_FLAGS=true \
VALIDATION_STRINGENCY=SILENT \
SO=coordinate \
ATTRIBUTES_TO_RETAIN=XS
|
4)Filter Consensus Reads
1
2
3
4
5
6
7
8
|
java -jar fgbio.jar FilterConsensusReads \
--input=test.consensus.merge.BAM \
--output=test.consensus.merge.filter.BAM \
--ref=reference --min-reads=2 \
--max-read-error-rate=0.05 \
--max-base-error-rate=0.1 \
--min-base-quality=30 \
--max-no-call-fraction=0.20
|
5)Clip
来源于同一个template的read,若果有重叠,重叠部分的突变其实应该只计一次,所以要clip一下read。
1
2
3
4
|
java -jar fgbio.jar ClipBAM \
--input=test.consensus.merge.filter.BAM \
--output=test.consensus.merge.filter.clip.BAM \
--ref=$ref --soft-clip=false --clip-overlapping-reads=true
|
三、Variant Call
1
2
3
4
5
6
|
AF_THR="0.01"
VarDict/bin/VarDict -G reference.fa -f $AF_THR -N test \
-b test.consensus.merge.filter.BAM \
-z -c 1 -S 2 -E 3 -g 4 -th 4 target.bed ' \
VarDictJava/VarDict/teststrandbias.R ' \
VarDictJava/VarDict/var2vcf_valid.pl -N test -E -f $AF_THR > test.vcf
|
参考:
https://github.com/fulcrumgenomics/fgbio
https://gatkforums.broadinstitute.org/gatk/discussion/6484/how-to-generate-an-unmapped-BAM-from-fastq-or-aligned-BAM
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5759201/
http://nilshomer.com/2017/07/05/single-strand-umi-somatic-variant-calling/
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5852328/
https://www.ncbi.nlm.nih.gov/pubmed/28100584
####################################################################
#版权所有 转载请告知 版权归作者所有 如有侵权 一经发现 必将追究其法律责任
#Author: Jason
####################################################################

