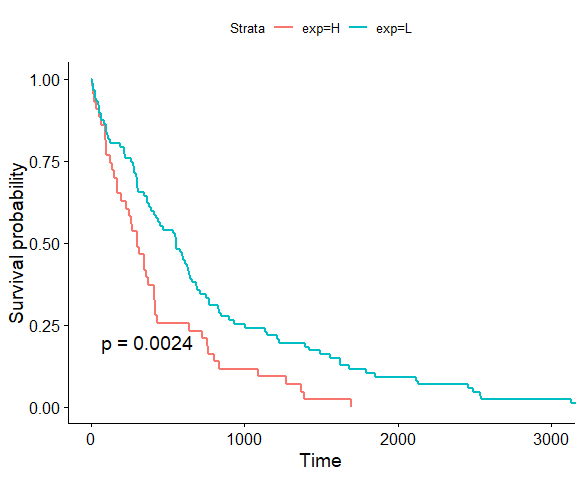
RefSeq的gtf文件
注释有很多版本,比如ensembl,gencode, ucsc known gene, NCBI的RefSeqGene。最近就需要NM id的注释,但NCBI提供的是gff3格式的,而且很乱。用UCSC table browser下载的gtf版本的RefSeq,没有转录本和基因之间的关系,也没有基因symbol。
比如Ensembl,其实Ensembl的gtf挺好用的,不过这次我因为需要NM编号的注释(笨方法是将ensembl id转成NCBI的refSeq的ID,但这不是最优的方法,ID mapping有可能对不上,不如直接用NM的注释)。
|
|
UCSC table browser下载的refGene的gtf,这个文件不对的地方是gene id和transcript id是一个,而我需要gene和transcript的关系
|
|