用Circos表示基因组上的突变密度
文章目录
不光是生信,感觉整个生物领域越来越靠图吃饭了。谁的图漂亮,谁的分析就好。好吧,我也承认,图多图漂亮了,确实显得高大上哈。在我眼中图的信息量比较大,能够给人直观的表示,为了说明图的意义,我没把持住,在本文多放了几张和circos图无关的图,见谅。本文有多图,都是本人自己画的,请勿盗图,如果你想知道怎么做或者由更好的表示办法,欢迎留言讨论。
想当年,我为了表示SNP在染色体上的数目分布,用python画了24个图,每个染色体一张SNP的密度分布。那可是我第一次用matplot。
先上我年yi轻qian时做的图,那个时候我都佩服自己能想出可以用图的形式来表示突变的密度,哈哈哈,因为这样就能很直观的看出哪些地方突变频率比较高,突变频率比较高的地方,可能是突变热点区域,在肿瘤研究中常常有意义。
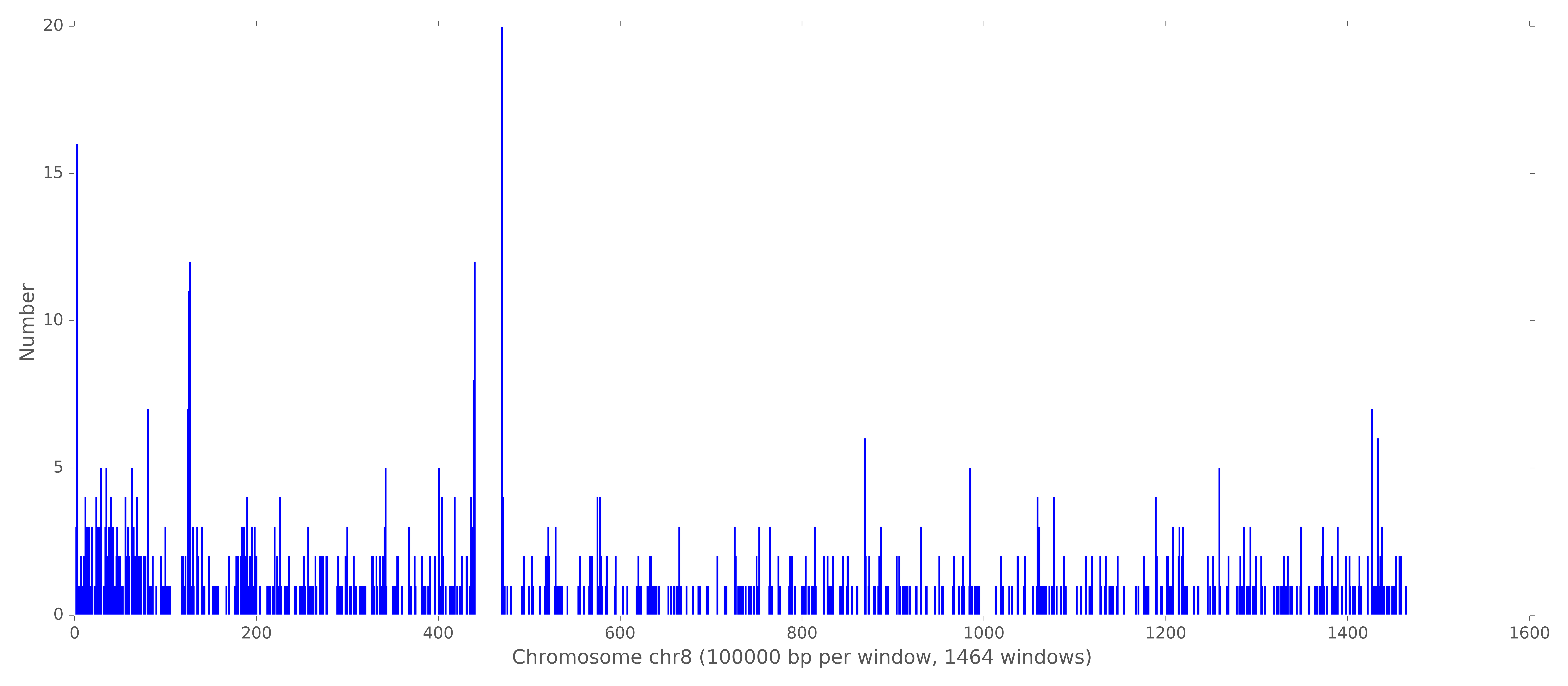
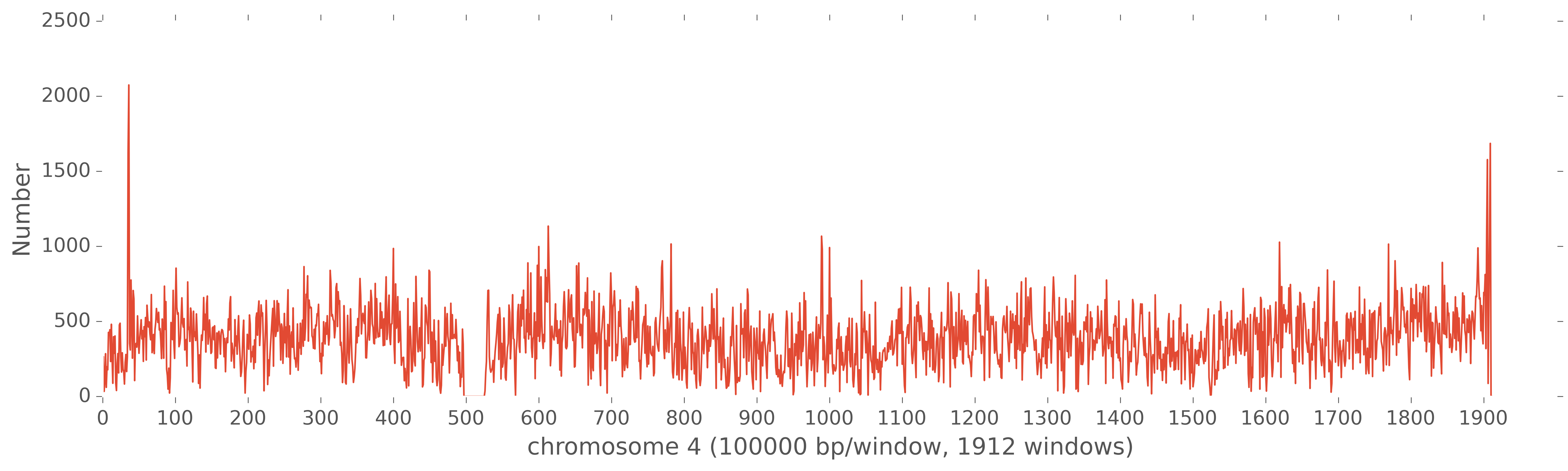
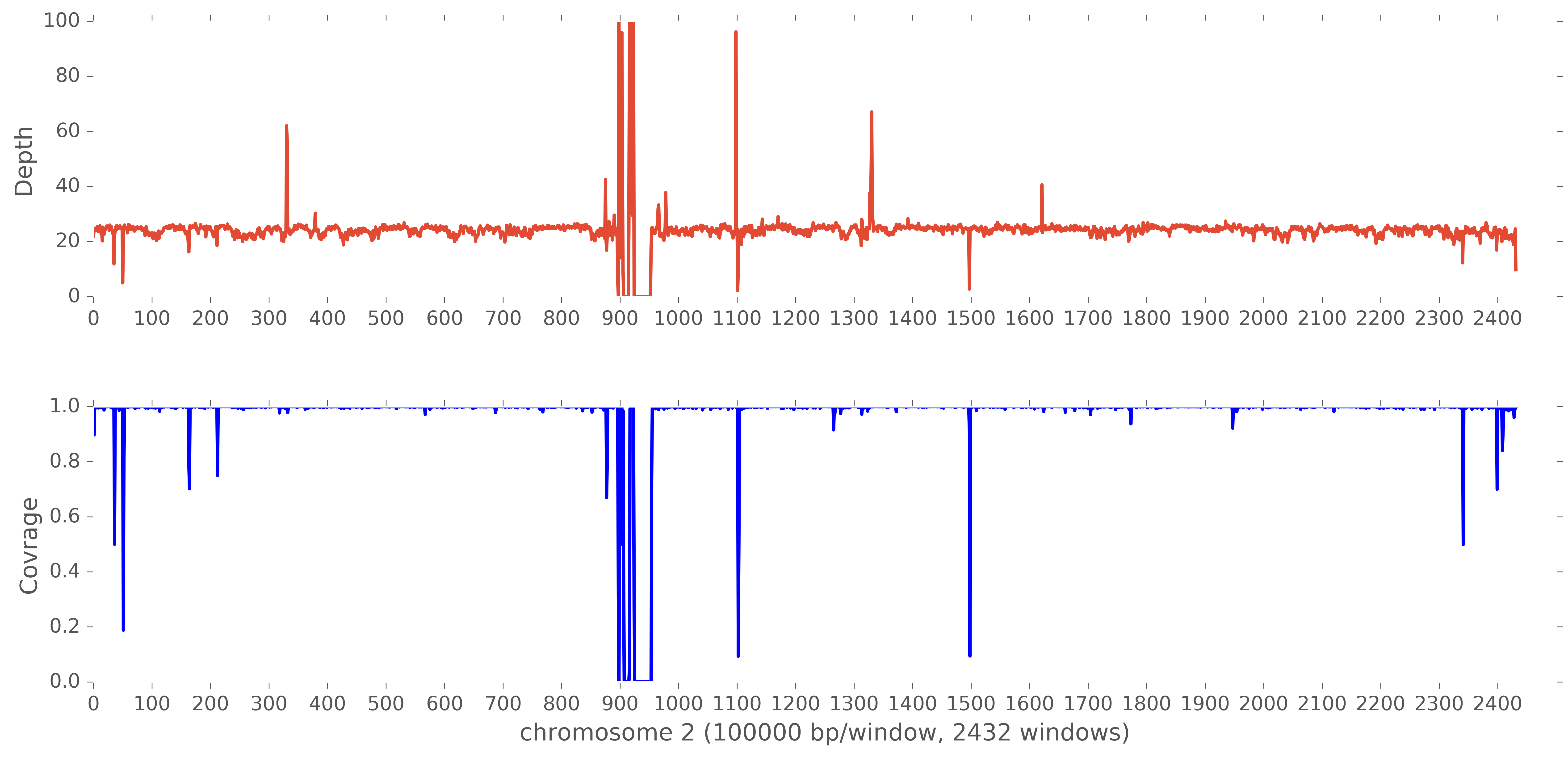
上面的两张图都比较丑,下面表示覆盖度和测序深度的图还算凑活。通过depth我们能够发现测序深度在整个染色体上比较均匀,为20X多。通过coverage我们可以看出,整条染色体的大部分都被reads覆盖到了。这种表示方法远比单纯告诉你测序深度多少要靠谱的多,你不仅能够看出染色体每个区域的测序深度,还能看到测序覆盖到的区域,很直观,不用列一堆数据在那,表示的信息量却相当丰富。这就是图的意义所在。
其实那个时候我就想到了可以用circos图表示,但是那个时候项目催的急,我很久之前帮一个师兄玩过一次circos图,不想那么麻烦了。最近越看越觉得丑,实在看不下去了,遂决定用circos来表示,顺便复习一下用circos作图。
circos我用的circos自带的测试数据,data/6/snp.number.1mb.txt。
我一共画了三圈,最外面的一圈,表示karyotype,包括染色体带,这个需要在配置中设置show_bands = yes,中间的一圈,用热图的形式表示每个区域内的突变的数目,第三圈用线来表示突变的数目,不过设置了orientation = in,让它朝内。突变的数目其实对应突变密度,只不过数量级不一样罢了。
上circos图,真心比我以前的图漂亮多了,而且是同一张图把整个基因组上的突变密度都表示出来了,非常直观漂亮。要是加上结构突变,覆盖度的信息,就更完美了,perfect!以后我会添上去的,嗯。
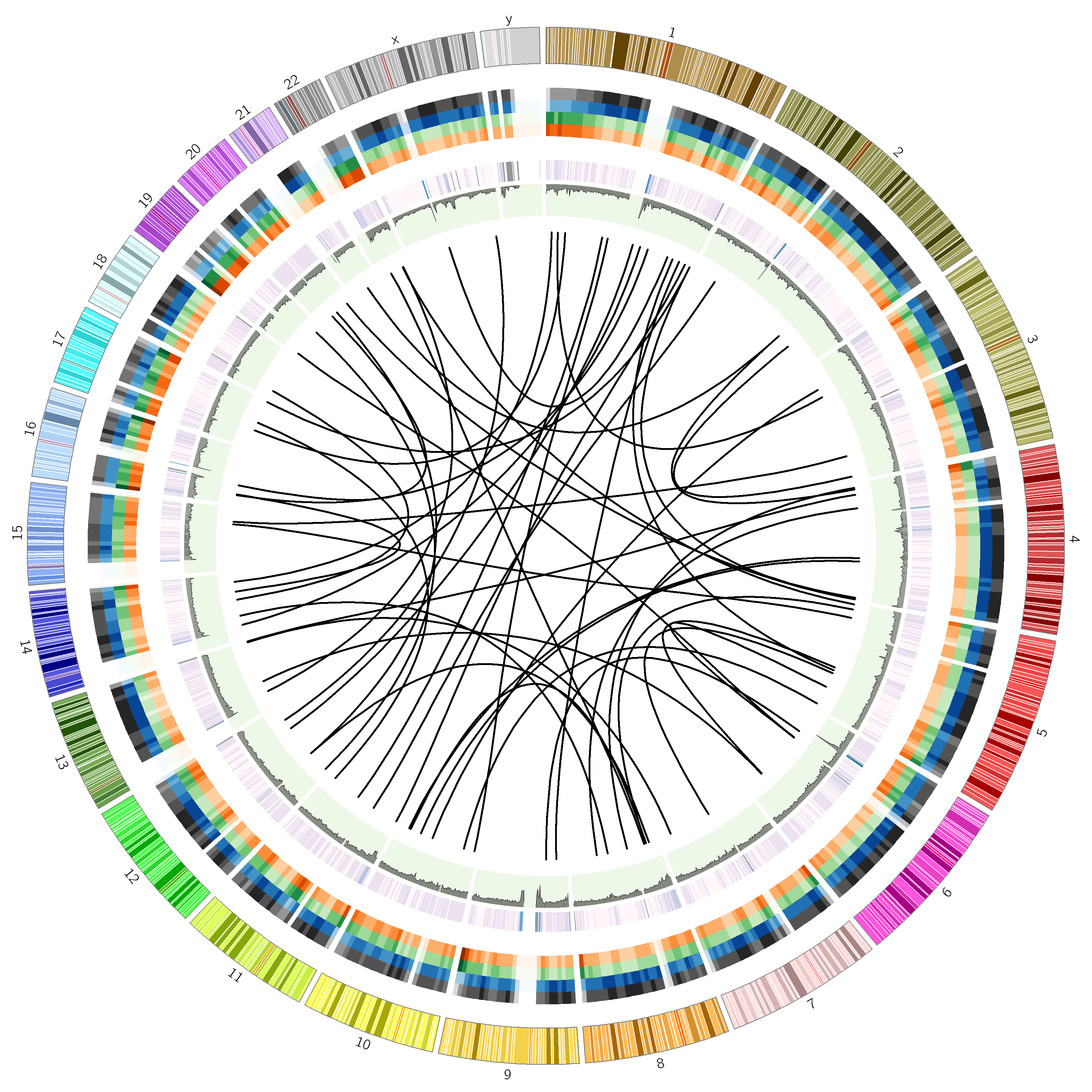
这里想提一下,各位有空可以看看circos作者提到的关于图片美学的论述,看后受益匪浅,不过我现在都忘了,囧。这也让我想到了ggplot2,这些搞艺术的,理论真强悍。人家画出的图就是漂亮,yes!
Circos图最大的优势是它以圈(track)的形式来表示,且可以画很多圈,能够表示的维度远比二维的要多。
下面的内容我主要介绍下如何在Windows下安装circos,以及circos的配置文件。
windows下安装circos
下载perl,安装 https://www.perl.org/get.html#win32
下载circos,解压缩 http://circos.ca/software/download
下一步需要安装其他的模块http://circos.ca/tutorials/lessons/configuration/perl_and_modules/
cd /path/to/circos/bin perl circos -modules # 检查缺失的模块 ok 1.36 Carp missing Clone missing Config::General ok 3.56 Cwd ok 2.158 Data::Dumper ok 2.54 Digest::MD5 ok 2.85 File::Basename ok 3.56 File::Spec::Functions ok 0.2304 File::Temp ok 1.51 FindBin missing Font::TTF::Font missing GD missing GD::Polyline ok 2.45 Getopt::Long ok 1.16 IO::File missing List::MoreUtils ok 1.41 List::Util missing Math::Bezier ok 1.9997 Math::BigFloat missing Math::Round missing Math::VecStat ok 1.03 Memoize ok 1.53 POSIX missing Params::Validate ok 1.64 Pod::Usage missing Readonly missing Regexp::Common missing SVG missing Set::IntSpan missing Statistics::Basic ok 2.53 Storable ok 1.20 Sys::Hostname ok 2.03 Text::Balanced missing Text::Format ok 1.9726 Time::HiRes 用PPM:进入安装的perl文件夹下的bin文件夹,右击ppm,选择perl打开 用cpan perl -MCPAN -e shell o conf urllist push http://mirrors.163.com/cpan/ o conf urllist push ftp://mirrors.sohu.com/CPAN/ o conf commit install Clone install Config::General install Font::TTF::Font install GD install GD::Polyline install List::MoreUtils install Math::Bezier install Math::Round install Math::VecStat install Params::Validate install Regexp::Common install Readonly install SVG install Set::IntSpan install Statistics::Basic install Text::Format
circos配置文件
####################configuration file###################
karyotype = test/karyotype.test.txt default = 0.005r ## http://circos.ca/documentation/tutorials/quick_start/ticks_and_labels/configuration # Ideogram position, fill and outline radius = 0.95r # 控制宽度 thickness = 50p fill = yes stroke_color = dgrey stroke_thickness = 2p # Minimum definition for ideogram labels. show_label = yes # see etc/fonts.conf for list of font names label_font = default label_radius = dims(image,radius) - 60p label_size = 30 label_parallel = yes # band 配置来自http://circos.ca/documentation/tutorials/2d_tracks/tiles/configuration show_bands = yes fill_bands = yes band_stroke_thickness = 2 band_stroke_color = white band_transparency = 4 ################################################################ # The remaining content is standard and required. It is imported # from default files in the Circos distribution. # # These should be present in every Circos configuration file and # overridden as required. To see the content of these files, # look in etc/ in the Circos distribution. # http://circos.ca/documentation/tutorials/2d_tracks/stacking_tracks/images # http://circos.ca/documentation/tutorials/2d_tracks/stacking_tracks/configuration show = yes type = heatmap color = spectral-5-div #stroke_thickness = 1 #stroke_color = black file = /data/6/snp.number.1mb.txt #内圈 r0 = 0.90r #外圈 r1 = 0.90r + 50p scale_log_base = 0.25 min = 0 # http://circos.ca/documentation/tutorials/2d_tracks/line_plots/configuration file = /data/6/snp.number.1mb.txt type = line max_gap = 1u color = black fill_color = black_a4 min = 0 # max = 0.015 r0 = 0.85r r1 = 0.85r + 60p thickness = 1 #fill_color = vdgrey_a3 # 朝里 orientation = in color = vvlgreen y0 = 0.006 color = vvlred y1 = 0.002 # Included from Circos distribution. <> # RGB/HSV color definitions, color lists, location of fonts, fill patterns. # Included from Circos distribution. <> # Debugging, I/O an dother system parameters # Included from Circos distribution. <>
####################configuration file###################
####################################################################
#版权所有 转载请告知 版权归作者所有 如有侵权 一经发现 必将追究其法律责任
#Author: Jason
###################################################################
文章作者 zzx
上次更新 2016-04-01
