Python抓取动态网页
文章目录
生物信息学中,David(the Database for Annotation,Visualization and Integrated Discovery)是常用的注视工具,可以对基因进行GO注释,KEGG pathway注释等,David提供接口供批量注释调用。
David的网址https://david.ncifcrf.gov/,api介绍https://david.ncifcrf.gov/content.jsp?file=DAVID_API.html
不是生物信息学的朋友,可以重点关注分析思路。 以Entrez gene id为1002的基因为例,返回GO,interpro等注释信息的api格式为
用python应用urllib2的包,抓取上述网页的结果为
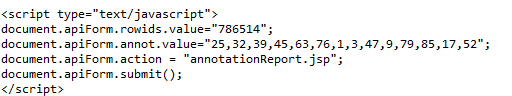
而不是直接点击连接的结果,这是为什么呢,因为这个网页是动态网页,是通过调用从数据库中得到数据,回显到网页上。但结果可以看到一个属性值,比如rowid.value应该对应的是基因,anno.value应该对应的是要返回的数据库,action给的是anntationReport.jsp,那么这个时候可以想到,应该是将这些值传给annotationReport.jsp的,这个jsp返回正确的结果。
如果在chrome下按F12,刷新网页,对网页连接过程进行分析,在annotationReport中的preview中看到了想要得到的信息。
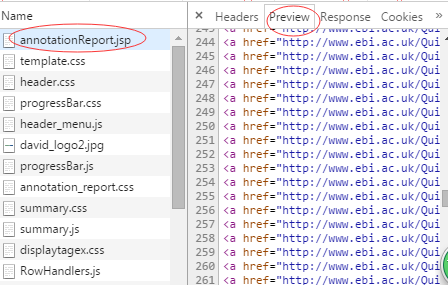
进一步分析其请求头信息,
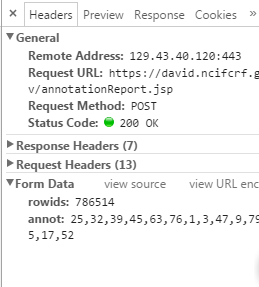
发现其用的是post,post的数据真是当初得到的rowid,anno信息。可以确定是这个annotationReport的jsp负责返回数据。
点击view url,得到传递的值,传递给annotationReport,构建URL
然后用python抓取,提示:
Your session has ended or you have removed all gene lists! Please go to the main DAVID page and upload a new list.
解决办法是,将请求头信息加到request中,要包括cookies,而且cookies有时效。重新请求,即可抓取id为1002的基因的注释信息。
如果你的网页刚开始看不出指向的jsp,可以按F12,看连接过程中,哪些文件的preview有你想得到的信息,并且在header中,post了你要关注的值,那么这个文件就是你要request的。
####################################################################
#版权所有 转载请告知 版权归作者所有 如有侵权 一经发现 必将追究其法律责任
#Author: Jason
####################################################################
文章作者 zzx
上次更新 2015-11-16
