**ELMER (Enhancer Linking by Methylation/Expression Relationships)**是基于甲基化数据和转录组数据,分析远端甲基化位点调控基因表达的R包,包里也整合了TCGA的数据,也可以自己构建MAE对象,快速进行分析,识别差异甲基化的位点,与基因表达显著相关的甲基化位点和相关区域的motif。
DNA 甲基化可用于识别肿瘤和其他原发性疾病组织中转录增强子和其他顺式调控模块(CRM,cis-regulatory modules)的功能变化。 R/Bioconductor 包 ELMER(通过甲基化/表达关系增强连接)提供了一种系统方法,通过结合来自同一组样本的甲基化和基因表达数据来重建基因调控网络 (GRN,gene regulatory networks)。 ELMER 使用 CRM 的甲基化变化作为网络调控的基础,利用相关性分析将它们与上游主调控 (MR,master regulator) 转录因子和下游目标基因相关联。
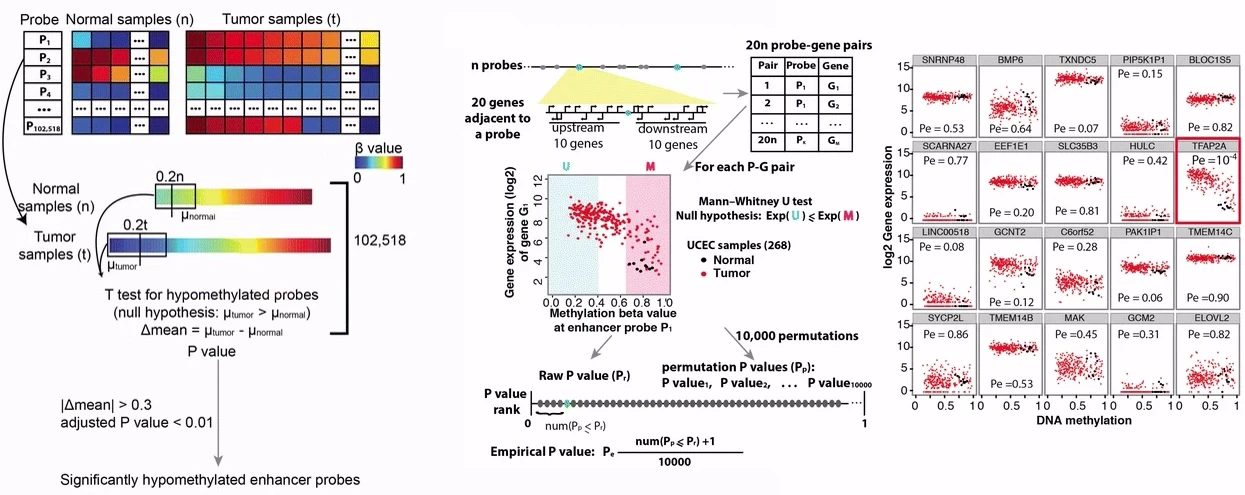
ELMER 分析有 5 个主要步骤:
- 识别 HM450K 或 EPIC 阵列上的远端probe(集成了注释,很方便的拉取远端probe的信息)
- 识别两组之间 DNA 甲基化水平显着不同的远端probe(常规的甲基化差异分析)
- 识别差异甲基化远端探针的假定靶基因(通关相关性分析,关联probe和gene)
- 识别远端探针的富集motif,这些基序具有显着差异甲基化并与假定的目标基因相关(识别motif)。
- 识别其表达与富集motif处的 DNA 甲基化相关的调节性 TF转录因子
ELMER能做的分析,参考手册的plot页面,例如https://www.bioconductor.org/packages/release/bioc/vignettes/ELMER/inst/doc/plots_scatter.html

