liftover,crossmap进行坐标转换时用到的chain文件介绍
文章目录
在做基因组坐标转换的时候,用crossmap和liftover的时候,会用到chain file。大家都在讲坐标转换会用到这个文件,却没有讲过chain文件的具体内容(百度和谷歌搜索结果都没有中文介绍,本文应该是第一个)。本文的内容都翻译自UCSC网站,原文https://genome.ucsc.edu/goldenpath/help/chain.html,希望能帮到大家了解这个文件。
UCSC chain文件 http://hgdownload.soe.ucsc.edu/goldenPath/hg38/liftOver/ Ensembl chain文件 https://sourceforge.net/projects/crossmap/files/Ensembl_chain_files/
chain file里面包含许多块alignment的信息(个人觉得可以理解为同源的地方,chain),其中每一块有一个header,记录alignment在两个版本中坐标,以及许多行alignment data line记录具体比对情况。
即每一块有 Header Line 和 Alignment Data Lines组成。形式如下,有两个chain
|
|
Header Lines
记录两个版本的序列对应区域
|
|
header line以chain开头 score – chain 值 tName – 染色体 (reference sequence) tSize – 染色体大小 (reference sequence) tStrand – 链strand (reference sequence) tStart – 比对开始 (reference sequence) tEnd – 比对结束 (reference sequence) qName – 检索染色体 (query sequence,对应的另外一个基因组版本 ) qSize – 检索染色体大小 (query sequence) qStrand – 链strand (query sequence) qStart – 比对开始 (query sequence) qEnd – 比对结束 (query sequence) id – chain ID
位置从0开始,前100bp的表示方法为0到100,接下的100bp为100到200,strand的值为-号时,对应的是反向互补序列。
Alignment Data Lines
Alignment Data Line有三列,记录区域内的具体对应信息,最后一行只有ungapped的序列长度。
|
|
size – ungapped(一致的区域)的长度 dt – 该block块(一致的区域)的结束位置,到下一个block快(下一个一致的区域)起始位置的长度,其实就是gapped block不一致的区域的长度 (reference sequence) dq – 该block块(一致的区域)的结束位置,到下一个block快(下一个一致的区域)起始位置的长度,其实就是gapped block不一致的区域的长度(query sequence)
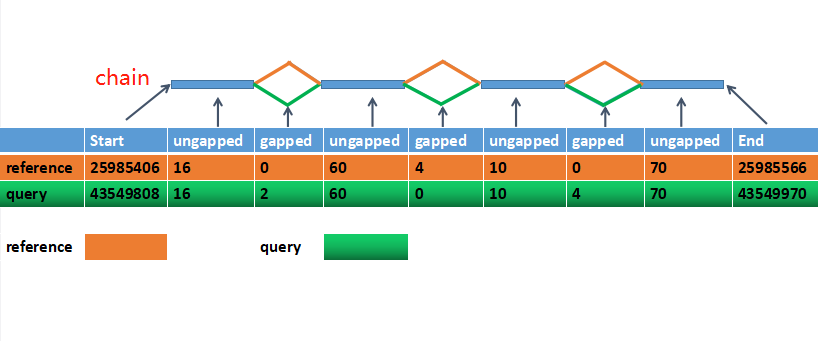
以chain id为2的chain为例
|
|
我个人理解的,不一定正确,欢迎指正

坐标转换在reference和query的位置为: reference: start 25985406 end 25985566 query: start 43549808 end 43549970
区域长度分别为: reference region length = 25985566 - 25985406 = 160 bp query region length = 43549970 - 43549808 = 162 bp
一致的区域(ungapped)长度为 reference = query = 16 + 60 +10 + 70 = 156 bp
不一致的区域(gapped)长度为 reference = 0 + 4 +0 =4 query = 2 + 0 + 4 =6
区域长度也可以通过ungapped和gapped区域计算,分别为: reference = ungapped + gapped = 156 + 4 =160 bp query = ungapped + gapped = 156 + 6 =162 bp
更多信息详见:
http://genomewiki.ucsc.edu/index.php/LiftOver_Howto
https://genome.ucsc.edu/goldenpath/help/chain.html
####################################################################
#版权所有 转载请告知 版权归作者所有 如有侵权 一经发现 必将追究其法律责任
#Author: Jason
####################################################################
文章作者 zzx
上次更新 2018-02-12
