基因组坐标转换工具-以BED文件为例,从hg19转换到hg38坐标
文章目录
分析时使用的基因组版本,可能会与其他来源数据所使用的基因组版本不一致,需要统一成同一个版本的坐标,才能方便下一步的分析。
常用的有NCBI的Remap在线服务和UCSC的liftover,其实还有很多,本文暂时总结部分工具的用法。以将APOA1的编码区坐标(利用UCSC的genome browser下载,或者下载该文件APOA1.bed)转换为例,从hg19转到hg38版本坐标上。需要注意的是,在使用的时候,需要注意是否支持对应的格式。
| 类型 | 支持格式 | 地址 | 推荐指数 | |
|---|---|---|---|---|
| Liftover | 在线 | bed | http://genome.ucsc.edu/cgi-bin/hgLiftOver | 一般 |
| Liftover | 本地 | bed和gff | http://hgdownload.cse.ucsc.edu/admin/exe/linux.x86_64/liftOver | 推荐 |
| Remap | 在线 | hgvs,bed,gvf,gff,gtf,Text ASN.1,Binary ASN.1,UCSC Region和VCF | https://www.ncbi.nlm.nih.gov/genome/tools/remap | 推荐 |
| CrossMap | 本地 | SAM/BAM,,Wiggle/BigWig, bed, gff/gtf,VCF | http://crossmap.sourceforge.net/ | 推荐 |
| picard | 本地 | interval和VCF | http://broadinstitute.github.io/picard/ |
在线版
Liftover
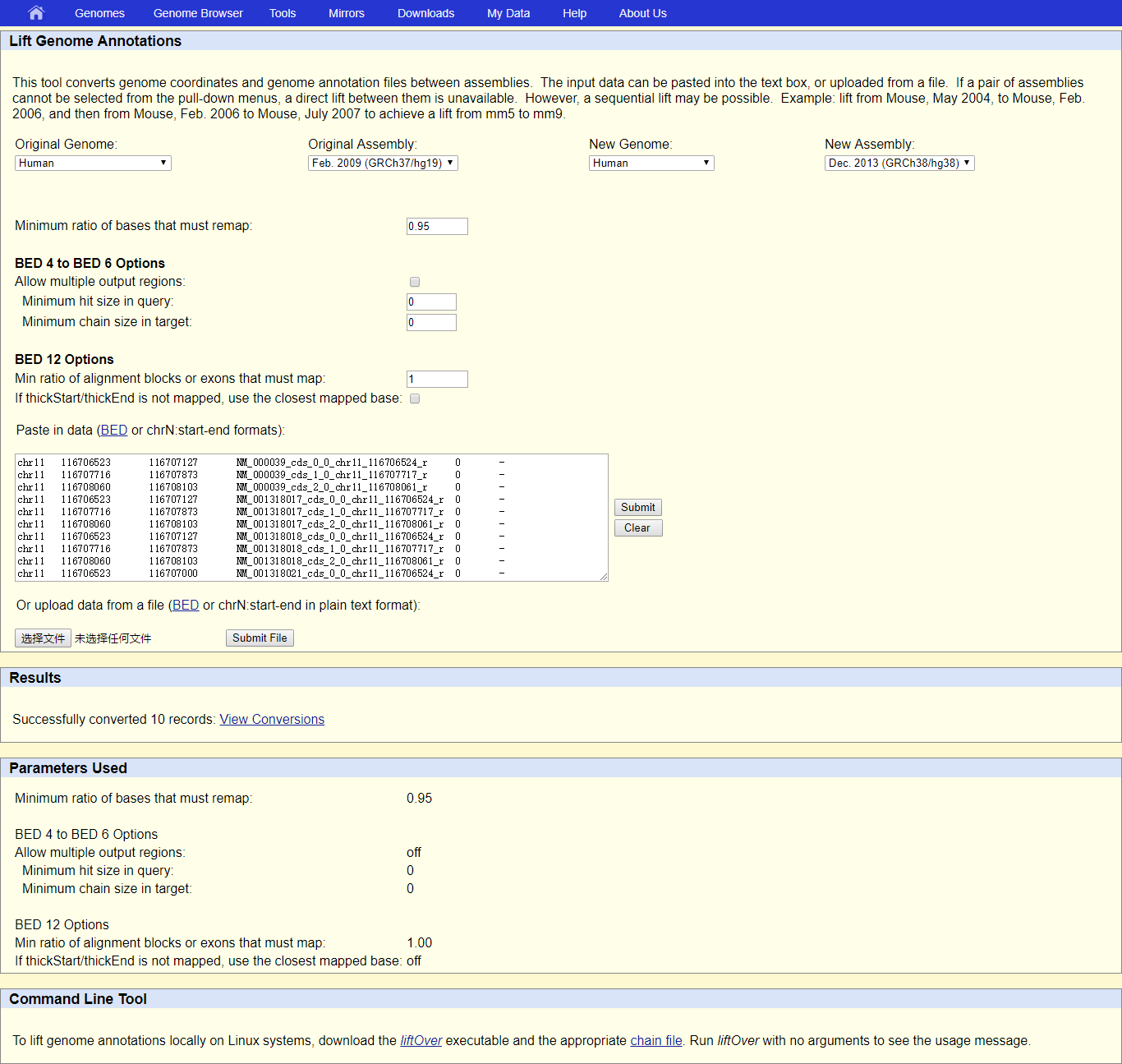
服务地址: http://genome.ucsc.edu/cgi-bin/hgLiftOver
-
- 1,首先选择检索和目标基因组
|
|
- 2,将bed格式内容复制或者上传,如果是复制内容的话,点击submit;如果是上传文件的 话,点击submit
- 3,在Result部分下载结果,Result部分会显示结果说明,点击View Conversions,即能得到转换后的bed文件。
Remap
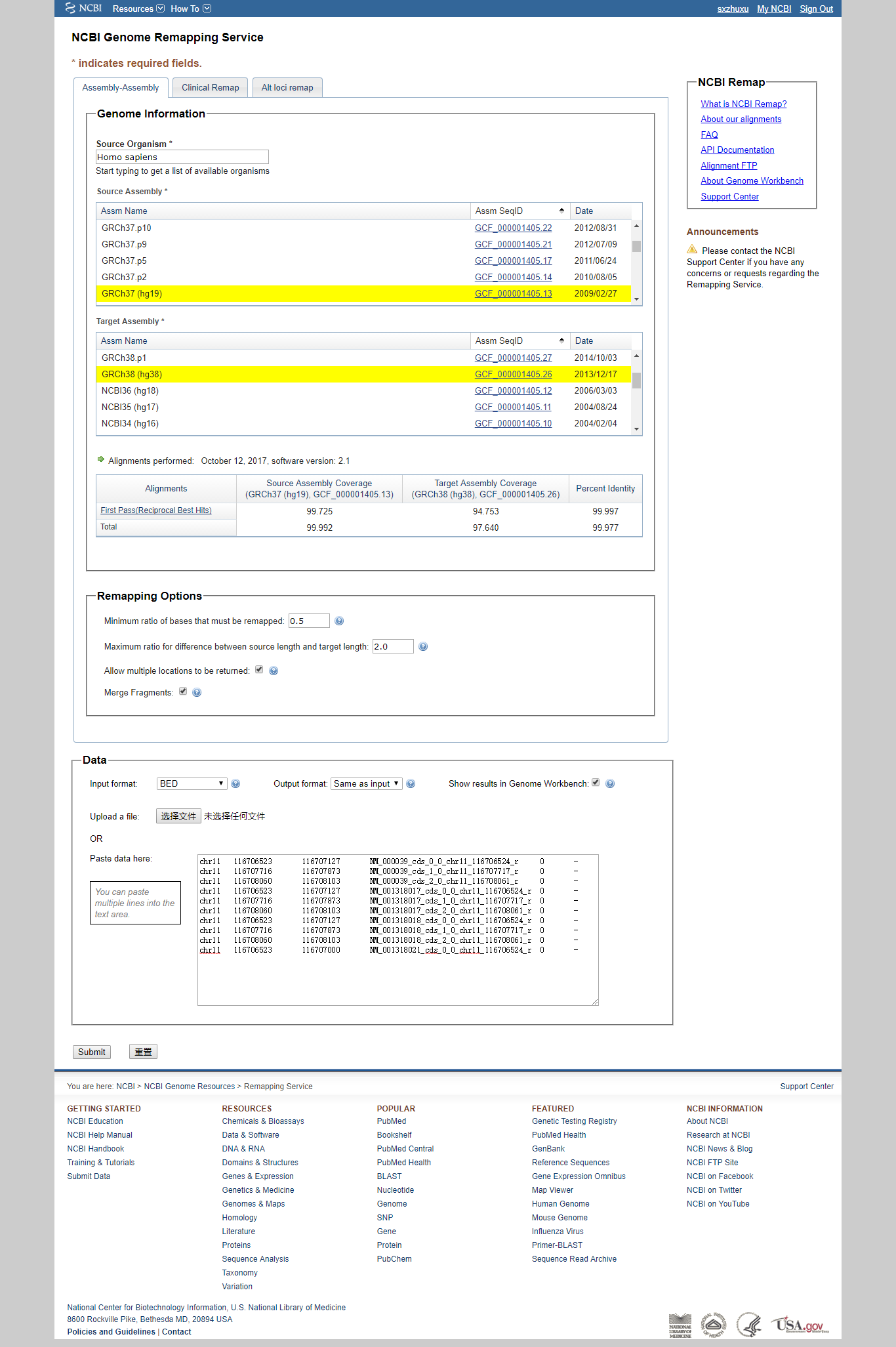
支持hgvs,bed,gvf,gff,gtf,Text ASN.1,Binary ASN.1,UCSC Region和VCF,如果数据量较少,可以考虑该方法。
服务地址:https://www.ncbi.nlm.nih.gov/genome/tools/remap
- 1,在Assembly-Assembly中选择
|
|
- 2,在Data处,可以选择复制文件内容还是上传文件,还可以指定文件格式。
- 3,点击submit,页面会跳转到结果页面,Summary Data告诉你有多少条匹配,Mapping Report告诉你对应关系。这些都可以下载,NCBI remap的结果文件前几列是原来的内容,后几列是转换后的坐标。
Ensembl Assembly Converter
http://asia.ensembl.org/Homo_sapiens/Tools/AssemblyConverter?db=core
本地版
本地工具需要利用chain file,才能知道两个版本的坐标对应关系。chain文件可以从UCSC下载。我们需要的是hg19Thg38.over.chain.gz http://hgdownload.soe.ucsc.edu/goldenPath/hg19/liftOver/hg19ToHg38.over.chain.gz chain文件后续会有介绍。
CrossMap
CrossMap支持SAM/BAM, Wiggle/BigWig, BED, GFF/GTF, VCF格式。支持的格式较多,用起来方便。
软件网址:http://crossmap.sourceforge.net/
安装:pip install CrossMap
当然也可以从源码编译安装,官网有说明(http://crossmap.sourceforge.net/#install-crossmap-from-source-code)。
|
|
Liftover
支持bed和gff格式
|
|
Picard
地址: http://broadinstitute.github.io/picard/
此外还有Picard可以完成interval和VCF的转换,但interval的转换比较繁琐,首先要构建picard风格的interval文件(参见https://gatkforums.broadinstitute.org/gatk/discussion/1319/collected-faqs-about-interval-lists),加入header,必须有 + 五列,第四列且是+号。所以不建议用picard来转换bed,不过可以用picard转换VCF文件。
|
|
####################################################################
#版权所有 转载请告知 版权归作者所有 如有侵权 一经发现 必将追究其法律责任
#Author: Jason
####################################################################
文章作者 zzx
上次更新 2018-02-11
