安装BioMart Perl及利用BioMart Perl API下载数据
文章目录
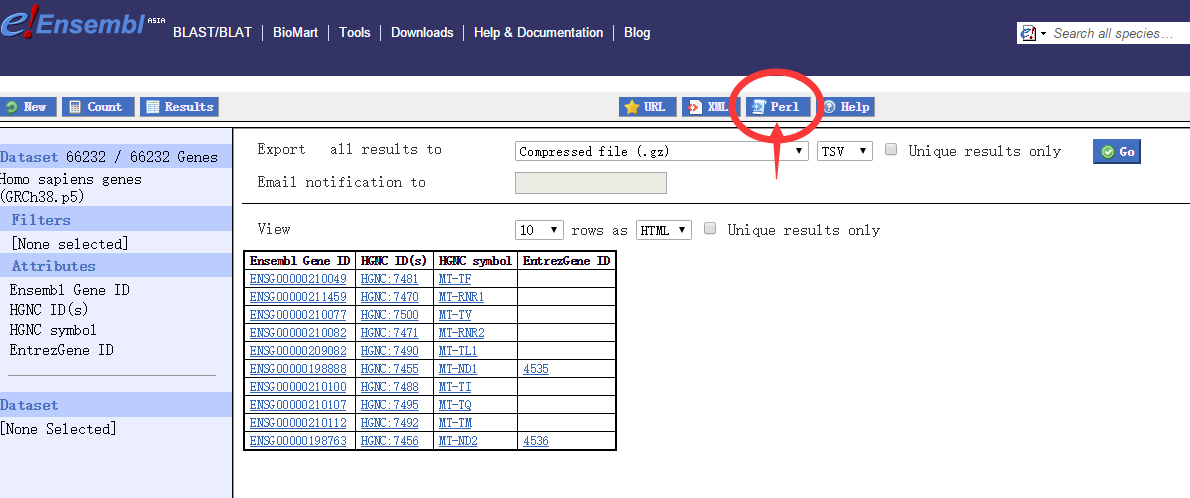
上篇介绍了如何利用ensembl的biomart服务下载ensembl gene id与NCBI entrez gene id的对应关系时,最后一步是保存result。biomart也提供通过biomart-perl,在本地通过perl脚本下载,并通过标准输出到终端上。biomart提供生成好的perl脚本,只需在选择好相关attribute和filter之后,点击中间上方的perl即可。
一,安装 biomart_perl
1,第一步就是安装biomart_perl
具体文档可以参考http://www.biomart.org/other/install-overview.html页面的1.2小节。
|
|
2,安装相关依赖模块
|
|
3,添加registry
通过添加registry,告诉biomart-perl相关的服务器地址。 编辑/path/to/biomart-perl/conf/martURLLocation.xml ,清除标签及标签中间的内容,打开http://asia.ensembl.org/biomart/martservice?type=registry
将此页面的内容复制到"$confFile"文件中。
二,准备biomart_perl脚本
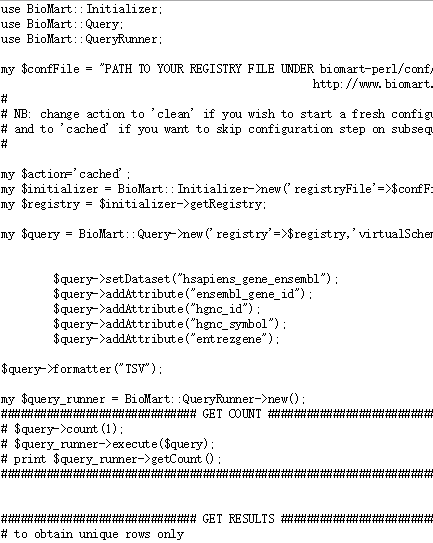
1,生成perl脚本
在选择好相关attribute和filter之后,点击中间上方的perl,保存生成的内容到本地,比如down.pl。
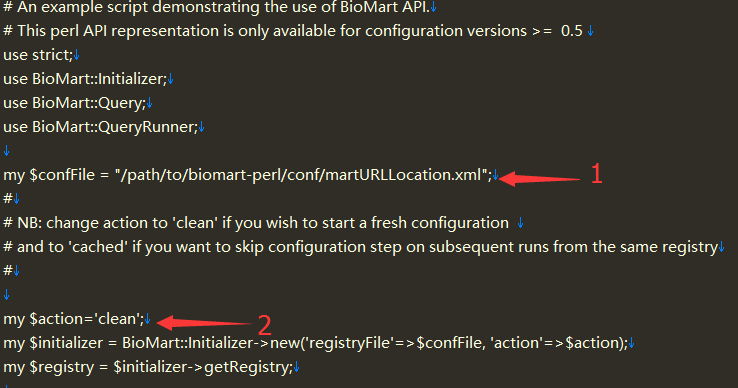
2,修改perl脚本
1) 将"$confFile" 变量换成本地的registry文件(比如,/path/to/biomart-perl/conf/martURLLocation.xml) 2) 将"$action" 变量改为"clean"
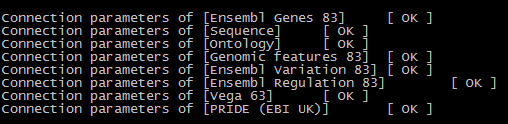
三,下载数据
perl downl.pl
参考:http://asia.ensembl.org/info/data/biomart/biomart_perl_api.html?redirect=no
####################################################################
#版权所有 转载请告知 版权归作者所有 如有侵权 一经发现 必将追究其法律责任
#Author: Jason
####################################################################
文章作者 zzx
上次更新 2016-02-19
